Участник:JuliaA/Алгоритм кластеризации с использованием представлений
Основные авторы описания: Ю.А. Антохина, С.А. Шатков
Содержание
- 1 ЧАСТЬ. Свойства и структура алгоритмов
- 1.1 Общее описание алгоритма
- 1.2 Математическое описание алгоритма
- 1.3 Шаг 1
- 1.4 Шаг 2
- 1.5 Вычислительное ядро алгоритма
- 1.6 Макроструктура алгоритма
- 1.7 Схема реализации последовательного алгоритма
- 1.8 Последовательная сложность алгоритма
- 1.9 Информационный граф
- 1.10 Ресурс параллелизма алгоритма
- 1.11 Входные и выходные данные алгоритма
- 1.12 Свойства алгоритма
- 2 ЧАСТЬ. Программная реализация алгоритма
- 3 Литература
1 ЧАСТЬ. Свойства и структура алгоритмов
1.1 Общее описание алгоритма
Пусть дан набор точек и заданы правила определения расстояния между ними. Задача кластеризации сгруппировать эти точки в определенное число кластеров. Члены одного кластера похожи, а члены разных классов различны.
В алгоритме CURE применяется иерархическая кластеризация. Есть два подхода к иерархической кластеризации. В одном случае один большой кластер на каждом шаге разбивается на несколько, пока число кластеров не будет равно заданному заранее числу кластеров. В другом случае наоборот кластеры, состоящие каждый из одной точки объединяются в большие. Второй метод используется в CURE.
Результат кластеризации зависит от того, как будут представлены кластеры. Например, средним всех точек кластера, или всеми точками кластера. Применение среднего возможно только в евклидовом пространстве. А использование всех точек неустойчиво к случайным выбросам. В CURE используется другой метод: для каждого кластера выбираются точки представители. Выбирается постоянное число точек, которые будут представлять каждый кластер. Кластеры с наиболее похожими наборами репрезентативных точек объединяются на каждом шаге алгоритма.
За счет использования точек представителей алгоритм CURE устойчив к выбросам и может выделять кластеры сложной формы и различных размеров.
Описание шагов
Шаг 1. Если все данные использовать сразу как входные для CURE, то эффективность алгоритма будет низкая, а время выполнения большим. Поэтому на первом шаге мы случайным образом выбираем часть точек которые помещаются в память, затем группируем наиболее похожие с помощью иерархического метода. Дальше работаем с кластерами.
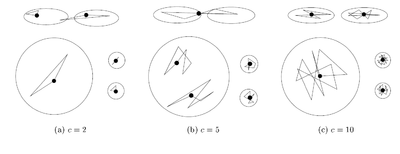
Шаг 2. Для каждого кластера выбираем c точек представителей, максимально удаленных друг от друга. Чисто c остается постоянным.
Шаг 3. Объединяем кластеры с наиболее похожими наборами точек представителей. Если не достигнуто нужное число кластеров, то перейти на шаг 2. Чтобы при объединении кластеров не выбирать каждый раз c точек представителей из всех точек, мы выбираем их только из 2c точек объединенных кластеров.
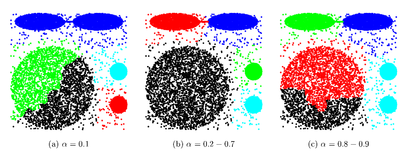
Выбранные точки сдвигаются на следующем шаге на \alpha к центроиду кластера. Алгоритм становится основанным на методе поиска центроида при \alpha = 1, и основанным на всех точках кластера при \alpha = 0.
1.2 Математическое описание алгоритма
Пусть дана матрица x \in R^{m\times n}, количество кластеров k, число точек представителей c, параметр \alpha, параметр \gamma. Каждая строка матрицы представляет собой точку в n - мерном пространстве.
m число точек для кластеризации, n размерность.
Расстояние \rho(p,k) между точками p = (p_{1},\dots,p_{n}) и k = (k_{1},\dots,k_{n}) будем вычислять по формуле:
\rho(p,k) = \sqrt{\sum_{i=1}^n (p_{i} - k_{i})^2}
Шаг "Инициализация"
Выбираются \lceil \gamma m\rceil строк матрицы случайным образом. Например, можно использовать Алгоритм R.
В основной памяти выделяется куча соответствующего размера для размещения дерева, листьями которого являются выбранные строки, соединённые ребром с вершиной дерева.
Шаг "Кластеризация"
В самом начале шага считаем каждую выбранную точку одноточечным кластером с центроидой в этой же точке.
Далее выполняем следующие шаги 1-2, пока количество кластеров не станет равно k:
1.3 Шаг 1
Для каждого i-го кластера вычисляется центроида \mu_{i} по формуле:
\mu_{i} = \frac{1}{s_{i}}\sum_{j\in S_{i}} x^{j}, где S_{i} - номера строк матрицы x, входящие в i-й кластер; s_{i} - количество элементов в множестве S_{i}; x^{j} - j-я строка матрицы x.
1.4 Шаг 2
Выполняется поиск пары кластеров, "наиболее подходящих" для объединения. В качестве критерия выбора можно рассматривать: наименьшее расстояние между центроидами кластеров или наименьшее расстояние между элементами, входящими в рассматриваемую пару кластеров. Существуют также другие критерии выбора "подходящих" классов, например, сравнение плотности кластеров до объединения и после. Далее выполняется слияние найденных кластеров. Для дерева это означает добавление новой вершины, соединённой ребром с корнем; поддеревья кластеров становятся ветвями этой новой вершины.
Шаг "Назначение представителей"
Шаг "Сдвиг представителей к центроиде"
Для каждого представителя выполняется сдвиг к центроиде соответствующего кластера в \alpha раз. (ПОЯСНЕНИЯ)
Шаг "Присваивание"
На данном шаге происходит присвоение неиспользованных точек (строк матрицы x) к кластерам. Для точки-строки p вычисляется расстояние до каждого представителя. Результат: строка p добавляется в поддерево кластера, которому принадлежит наиближайший представитель.
1.5 Вычислительное ядро алгоритма
1.6 Макроструктура алгоритма
1.7 Схема реализации последовательного алгоритма
1.8 Последовательная сложность алгоритма
Максимальное время выполнения алгоритма O(n^2 logn). Для двумерного пространства временная сложность может быть O(n^2). Так что сложность алгоритма не хуже чем у центроидного. То есть при такой размерности входных данных CURE выполняется за полиномиальное время.
(ПОЯСНЕНИЯ, ЕСЛИ ЕСТЬ ВРЕМЯ)
1.9 Информационный граф
1.10 Ресурс параллелизма алгоритма
1.11 Входные и выходные данные алгоритма
Входные данные: матрица x \in R^{m\times n}, количество кластеров k, число точек представителей c, параметр \alpha, параметр \gamma.
m число точек для кластеризации, n размерность.
Параметр \alpha для сжатия точек представления к центру кластера. В оригинальной статье авторы показывают что \alpha от 0.2 до 0.7 помогает хорошо выявлять несферические кластеры и подавлять редкие выбросы.
Параметр \gamma задаёт какая часть от m точек будет использована при случайной выборке точек на шаге "Инициализация".
Число точек представителей выбирается в зависимости от данных. Если возможно появление кластеров сложной формы, то точек должно быть больше, чтобы выявить их форму. В оригинальной статье авторы показывают что уже при c равном 10 достигается неплохая эффективность.
1.12 Свойства алгоритма
Алгоритм CURE выполняет иерархическую кластеризацию с использованием набора определяющих точек. Он действует только в евклидовом пространстве и предназначен для кластеризации очень больших наборов числовых данных.
Преимущества: выполняет кластеризацию на высоком уровне даже при наличии выбросов, выделяет кластеры сложной формы и различных размеров, обладает линейно зависимыми требованиями к месту хранения данных и квадратичную временную сложность для данных высокой размерности.
Недостатки: есть необходимость в задании пороговых значений и количества кластеров, работает только на числовых данных, эффективен только для данных низкой размерности
2 ЧАСТЬ. Программная реализация алгоритма
2.1 Масштабируемость алгоритма и его реализации
Данный раздел будет заполнен к 15 ноября.
2.2 Существующие реализации алгоритма
Последовательная реализация CURE-алгоритма для кластеризации для Python/C++ представлена в пакете: pyclustering.
Также существует параллельная реализация алгоритма, описанная в сборнике [3]. Алгоритм предложенный в статье использует массив данных состоящий из размеров кластера, его центроида и репрезентативных точек, а при объединении кластеров информацию о новом кластере просто записывают в строчку предыдущего. Также для каждого алгоритма требуется считать индекс и расстояние до ближайшего кластера. Именно их поиск авторы статьи и предлагают распараллелить.
3 Литература
[1] Воеводин В.В., Воеводин Вл.В. Параллельные вычисления. - СПб.: БХВ-Петербург, 2002. - 608 с.
[2] Sudipto Guha (Stanford), Rajeev Rastogi (Bell Labs), Kyuseok Shim (Korea Institute of Technology). CURE: an efficient clustering algorithm for large databases. SIGMOD '98 Proceedings of the 1998 ACM SIGMOD international conference on Management of data, pp 73-84
[3] Panagiotis E. Hadjidoukas, Laurent Amsaleg. OpenMP Shared Memory Parallel Programming. Volume 4315 of the series Lecture Notes in Computer Science pp 289-299 Parallelization of a Hierarchical Data Clustering Algorithm Using OpenMP